バイオ事業 プロテオーム解析支援
Sequence Composer
関係論文
Takao Kawakami, Junko Ozaki, kazuhiro Kondo, Shinji Sato and Harunoru Yunokawa.
Amino Acid Sequence Database Suitable for the Protein and Proteome Analysis.
Current Proteomics, 2008, 5, 267-274
Sequence Composerとは?
- ペプチド/プロテインの同定精度向上を実現する成熟蛋白質構造と多様性を考慮したアミノ酸配列DB作成ソフトウェアです。
- 公共の蛋白質データベース(UNIPROTやNCBI Nr)のアミノ酸配列構造は、一般的に蛋白質の全長配列で構成されています。しかしながら、質量分析装置で取得されたスペクトラのもととなるペプチド/プロテインは、多様性や成熟蛋白質の構造をもっている場合があります。Sequence Composerは、1アミノ酸配列に付随するアノテーションを利用し、考えうる全てのアノテーションの組み合わせを反映させたアミノ酸配列を生成しDB化します。本ソフトウェアで作成したデータベースを用いることで質量分析法による蛋白質同定精度の向上を実現します。
主な特徴
- 質量分析装置で取得したスペクトラとペプチド/プロテインを照合させるアルゴリズムに、本ソフトウェアで作成した配列データを用いると、低いスコアで同定されたペプチド/プロテインの同定スコアの向上を図ることができます。
- また、照合結果に含まれる擬陰性の疑いを見極めるアミノ酸配列構造の確認作業に費やす時間や労力の大幅な低減を図ることができます。
- 本ソフトウェアは、PC上で動作し操作性の良いGUIによりフレキシブルに利用目的の配列データを作成することができます。
機 能
多様性や成熟蛋白質の構造を考慮したアミノ酸配列を生成しデータベース化
1アミノ酸配列に付随する、遺伝的な多様性、転写産物の多様性、蛋白質の加工・修飾の全ての組み合わせを考慮したアミノ酸配列データを生成しデータベース化します。
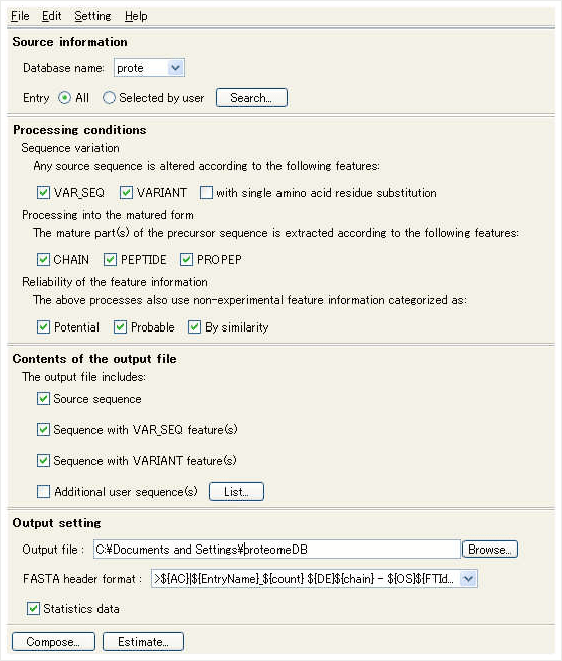
加工対象のコンディションを選択
ダウンロードしたUNIPROTのフラットファイルにアノテーションとして付随する情報をもとにした下記の加工条件を選択することができます。
VAR_SEQ(Variants from the DNA sequence)
VARIANT(Variants from the alternative splicing events)
CHAIN,PEPTIDE,PROPEP=Matured Form(Main chain processing)
VAR_SEQ(Variants from the DNA sequence)
VARIANT(Variants from the alternative splicing events)
CHAIN,PEPTIDE,PROPEP=Matured Form(Main chain processing)

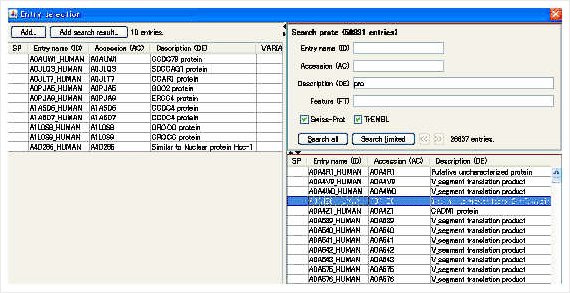
対象アミノ酸配列をDescriptionやAccession等で絞り込こみ可能
ユーザが、加工したい対象の蛋白質をDescriptionやAccessionで、検索し選択することができます。また、選択する際に指定した蛋白質のFeature情報も参照することができます。

加工後の配列数を確認可能
加工し生成したアミノ酸配列数の総数を確認することができます(統計情報の機能拡張計画中)。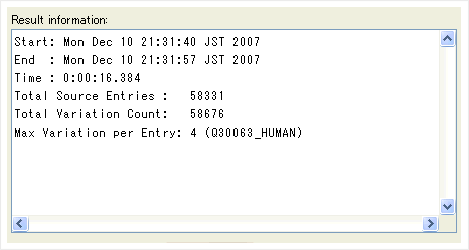

数十秒〜数分でプロセッシング可能
uniprot_sprot_human.datをプロセッシングし、データベース化が完了するまでの所要時間は約47秒です。(メモリ1GBのPCを利用)
☆ 東京医科大学川上隆雄先生との共同研究☆ 第55回 米国質量分析学会年会(55th ASMS)にてポスター発表
☆ 特許出願中
☆ 本製品は、東京都中小企業振興公社助成事業により製品化しました
出力結果
ペプチドの同定精製度向上
同じLC-MS/MSの結果に対して、2種類のアミノ酸配列DBを使って、検索エンジンで検索した結果、公共のアミノ酸配列DB(X軸)では高いスコアをえられなかった25個のペプチドに対して、成熟構造と多様性を考慮したアミノ酸配列DB(Y軸)では、高いスコアを得ることができた。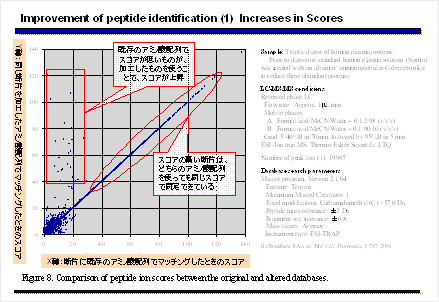

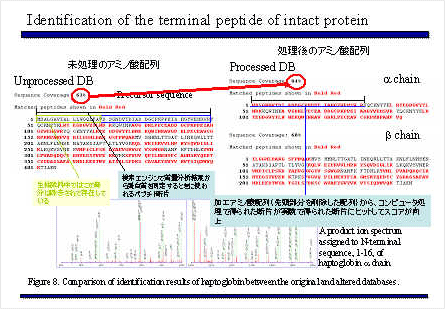
プロセッシング後のアミノ酸配列構造
シグナルペプチドを削除したアミノ酸配列を使うと、N末端のペプチドにヒットし、データベースのアノテーションを精査することなくα鎖が同定できていることがわかる。また、α鎖のみの配列にしたことで、スコアもアップした。